学术中心
PlantSeg:用于植物病害分割的大规模野外数据集
发布时间:
2024-10-16
来源:
作者:
植物病害对农业构成重大威胁。它需要正确的诊断和有效的治疗,以保障作物产量。为了实现诊断过程的自动化,通常采用图像分割技术来精确识别病害区域,从而推进精准农业的发展。开发稳健的植物病害图像分割模型需要在大量图像中提供高质量的标注。然而,现有的植物病害数据集通常缺乏分割标签,并且往往局限于受控的实验室环境,不能充分反映自然环境的复杂性。基于这一事实,我们建立了一个大规模的植物病害分割数据集PlantSeg。PlantSeg 与现有数据集的区别主要体现在三个方面。(1) 注释类型:与大多数仅包含类标签或边界框的现有数据集不同,PlantSeg 中的每张图像都包含详细、高质量的分割掩码,并与植物类型和疾病名称相关联。(2) 图像来源:与包含实验室环境图像的典型数据集不同,PlantSeg 主要包含野外植物病害图像。这种选择增强了实际应用性,因为训练有素的模型可用于综合病害管理。(3) 规模:PlantSeg 范围广泛,包含 11,400 幅带有病害分割掩膜的图像和另外 8,000 幅按植物类型分类的健康植物图像。大量的技术实验验证了 PlantSeg 注释的高质量。该数据集不仅能让研究人员评估其图像分类方法,还为开发先进的植物病害分割算法并为其设定基准奠定了重要基础。
 图1 PlantVillage 和我们的数据集的图像示例。由于是在实验室环境中收集的,PlantVillage 中的每张图片只包含一片叶子,并且背景统一,而我们数据集中的图片则具有更复杂的背景、不同的视角和不同的光照条件。
图1 PlantVillage 和我们的数据集的图像示例。由于是在实验室环境中收集的,PlantVillage 中的每张图片只包含一片叶子,并且背景统一,而我们数据集中的图片则具有更复杂的背景、不同的视角和不同的光照条件。
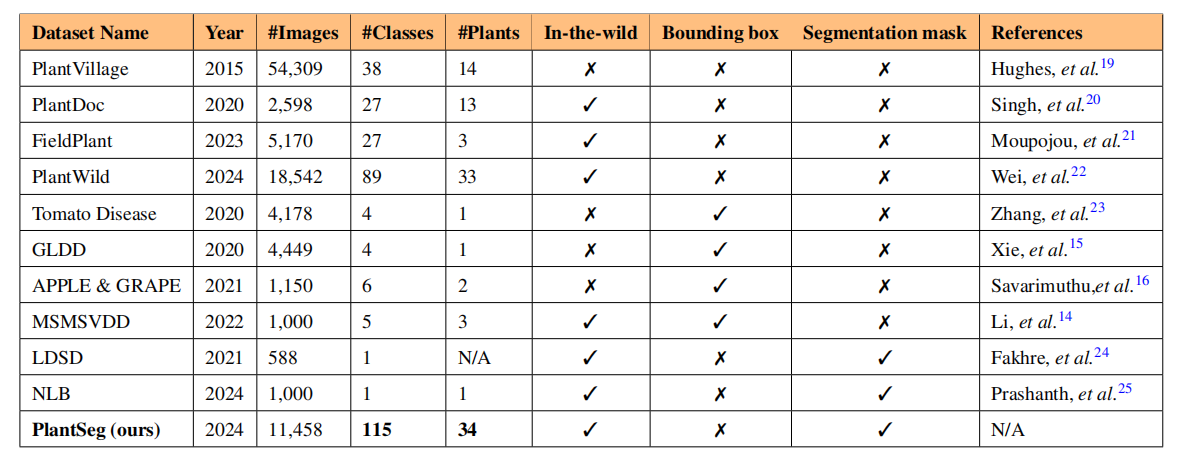 表1 植物病害图像数据集摘要。现有数据集
表1 植物病害图像数据集摘要。现有数据集
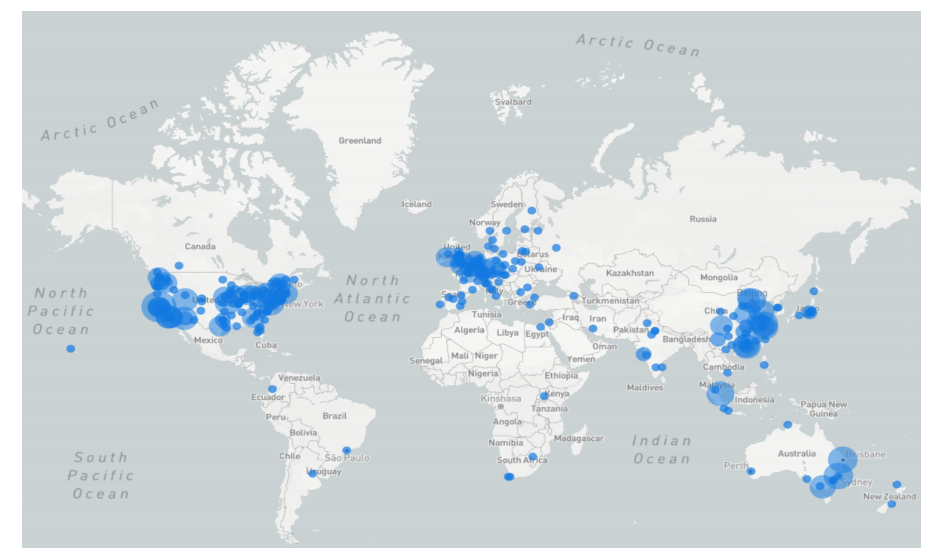 图2 获取的源图像的位置。图的大小表示获取图像的数量。每个圆圈的大小表示从该地址获取的图像数量,颜色深度表示附近区域内的地址密度。
图2 获取的源图像的位置。图的大小表示获取图像的数量。每个圆圈的大小表示从该地址获取的图像数量,颜色深度表示附近区域内的地址密度。
 图3 在受疾病影响的区域标注多边形的图像示例。
图3 在受疾病影响的区域标注多边形的图像示例。
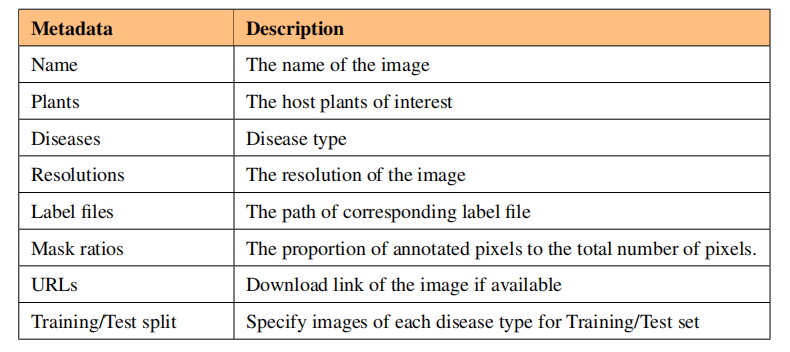 表2 PlantSeg 的元数据。
表2 PlantSeg 的元数据。
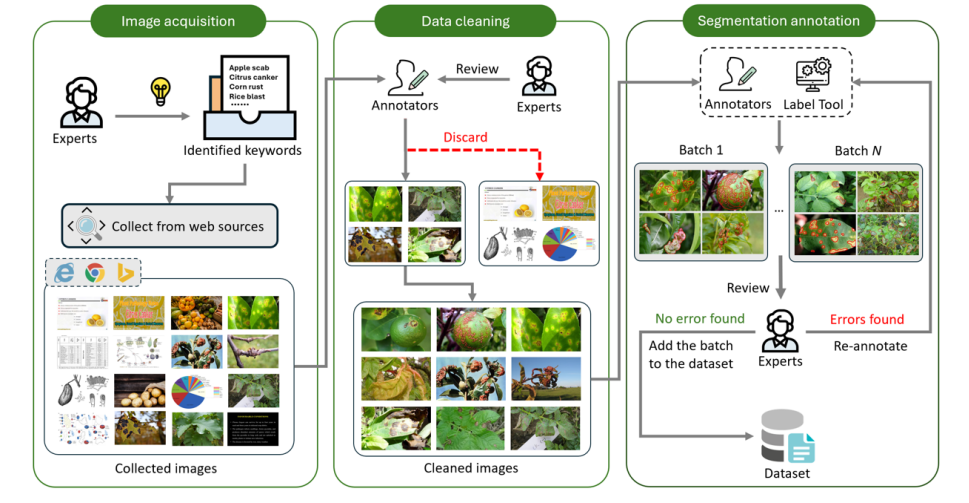 图4 PlantSeg 数据集的整理过程包括三个主要步骤:图像采集、数据清理和注释。在图像采集阶段,使用已识别的关键字从各种互联网资源中收集图像,然后根据其类别进行存储。在数据清理阶段,要识别并删除不正确的图像。在分割注释过程中,注释者利用 LabelMe26 对清理后的图像进行注释。这些注释随后由专家进行审核,并保存在 JSON 文件中。
图4 PlantSeg 数据集的整理过程包括三个主要步骤:图像采集、数据清理和注释。在图像采集阶段,使用已识别的关键字从各种互联网资源中收集图像,然后根据其类别进行存储。在数据清理阶段,要识别并删除不正确的图像。在分割注释过程中,注释者利用 LabelMe26 对清理后的图像进行注释。这些注释随后由专家进行审核,并保存在 JSON 文件中。
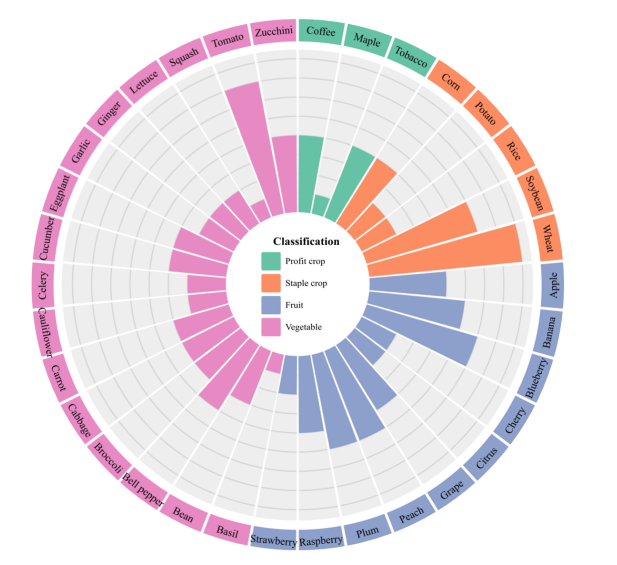 图5 PlantSeg 中的疾病分布根据植物和社会经济分类。条形的高度代表与每种植物相关的疾病数量。
图5 PlantSeg 中的疾病分布根据植物和社会经济分类。条形的高度代表与每种植物相关的疾病数量。
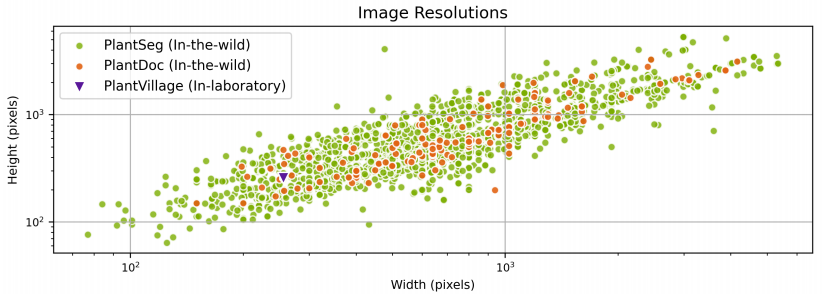 图6 不同数据集(包括 PlantVillage、PlantDoc 和 PlantSeg)的分辨率分布。每个绿点代表 PlantSeg 数据库中的一幅图像;红色代表 PlantDoc 数据库中的每幅图像;倒置的黄色三角形代表 PlantVillage 数据库。
图6 不同数据集(包括 PlantVillage、PlantDoc 和 PlantSeg)的分辨率分布。每个绿点代表 PlantSeg 数据库中的一幅图像;红色代表 PlantDoc 数据库中的每幅图像;倒置的黄色三角形代表 PlantVillage 数据库。
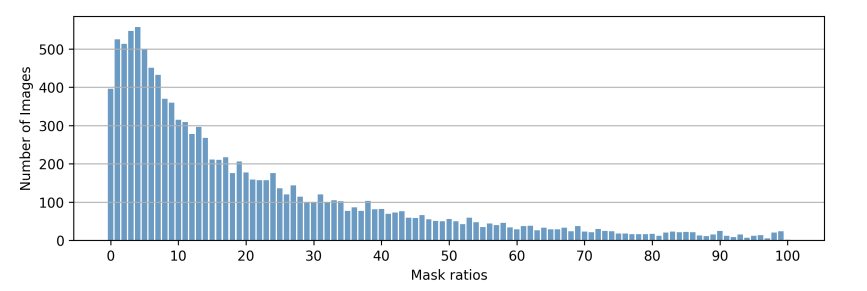 图7 横轴表示掩模区域相对于整个图像的百分比,纵轴表示对应图像的数量。
图7 横轴表示掩模区域相对于整个图像的百分比,纵轴表示对应图像的数量。
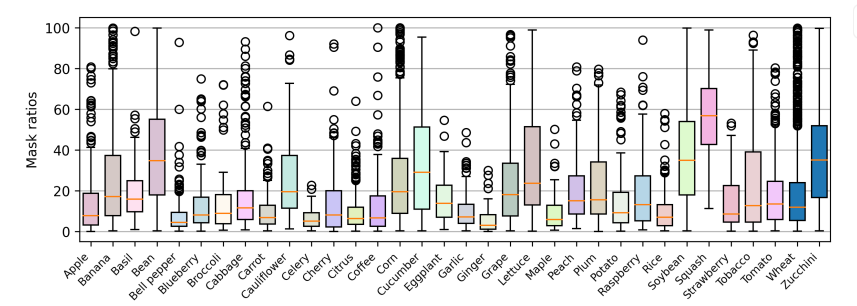 图8 箱线图显示了每幅图像掩模区域的百分比分布,不同植物之间的差异很大。
图8 箱线图显示了每幅图像掩模区域的百分比分布,不同植物之间的差异很大。
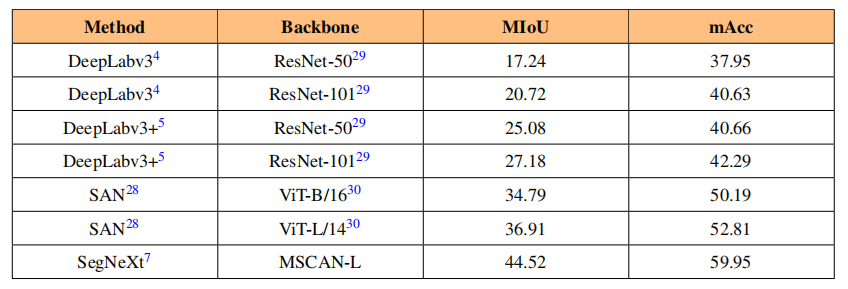 表3 PlantSeg 上不同方法的性能比较。
表3 PlantSeg 上不同方法的性能比较。
 图9 PlantSeg 测试集上的一些实验结果可视化。从左到右:PlantSeg 的图像示例、DeepLabv3的结果、SegNext的结果以及地面实况注释。
图9 PlantSeg 测试集上的一些实验结果可视化。从左到右:PlantSeg 的图像示例、DeepLabv3的结果、SegNext的结果以及地面实况注释。
Wei, T., Chen, Z., Yu, X., Chapman, S., Melloy, P., & Huang, Z. (2024). PlantSeg: A Large-Scale In-the-wild Dataset for Plant Disease Segmentation. arXiv.
编辑
小安
推荐新闻
视频展示
